检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
近日,学院林树海教授团队在Nature Communications、Chinese Chemical Letters和Analytical Chemistry发表三篇研究论文。研究团队通过创新性融合人工智能算法与质谱分析技术,在脂质组学注释效率、心磷脂结构解析及代谢组学定量分析等方向实现重要技术跨越,为精准医学和生物医学研究提供了系列重要分析工具。
近年来,液质联用技术(LC-MS)已成为定量脂质组研究的主要方法,但在注释准确率、搜库效率及深度结构解析等方面仍存在显著瓶颈。2025年5月16日,林树海教授团队在Nature Communications上发表题为LipidIN: a comprehensive repository for flash platform-independent annotation and reverse lipidomics的研究论文,推出首个快速、平台无依赖的反向脂质组学质谱AI模型LipidIN,显著提升脂质组鉴定速度与深度。

该研究首次将保留时间群智能(lipid categories intelligence,LCI)模型与反向脂质组学(Wide-spectrum Modeling Yield network,WMYn)模型引入脂质质谱数据分析,开发出全新的分析软件LipidIN(图1)。该平台通过构建涵盖分子链组成与碳碳双键位置信息的1.685亿条脂质碎片的分层谱库,并采用优化索引算法实现每秒超1000亿次查询。基于保留时间规律开发的脂质群模型(LCI)将错误发现率控制在5.7%,成功注释跨物种的8923种脂质,同时整合谱图再生成网络(WMYn)通过深度学习重构脂质碎片特征图谱,使目标分子召回率提升20%。在临床队列研究中,LipidIN已成功应用于脂质注释与生物标志物发现,其标准化分析流程显著提升了跨平台数据的可比性与可重复性。
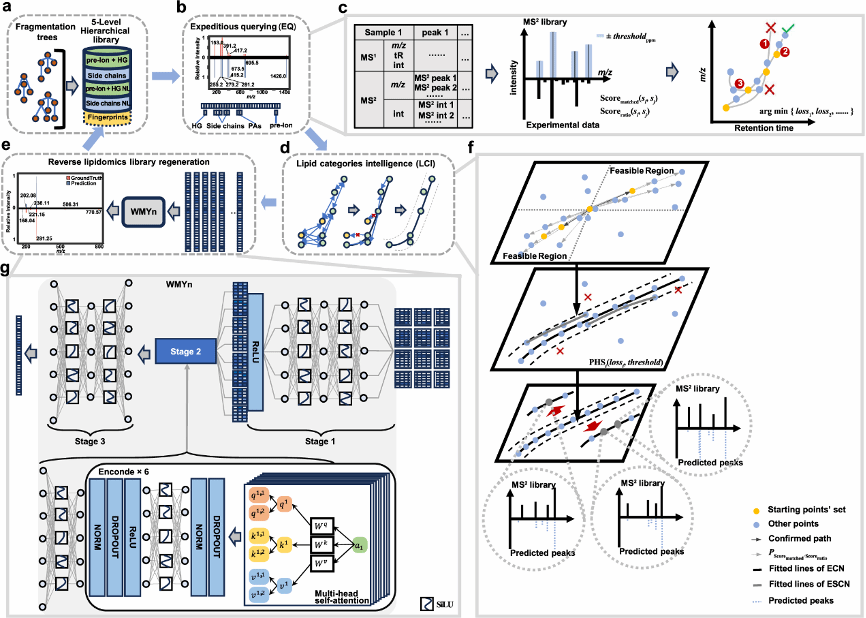
图1 LipidIN计算框架
该研究对LipidIN的性能进行了全面评估,并将其与现有的分析工具MS-DIAL,LipidSearch,Spectral Entropy,LipidMatch进行了比较。在注释速度测试中,采用低内存单线程计算机对公开数据集(MetabolomicsWorkbench ST001794)进行验证,EQ算法在千万级谱库中保持1000万次搜库仅需2.3微秒的稳定速度,而Flash Entropy在百万级谱库下单次MS2匹配耗时增至0.14秒。通过哈希表与二分法联合优化,EQ算法实现十亿次MS2谱图比对仅需0.23毫秒(每秒超4万亿次),较Flash Entropy提速6万倍(图2)。注释覆盖度测试显示,EQ模块联合MS-DIAL公共数据库在Top-20候选列表中召回率达70%,而整合LCI模块的四级分层谱库策略将召回率提升至90%以上。针对假阳性控制,LCI模块通过ECN/IUP/ESCN三重保留时间规则建立动态校正模型,在多数脂质亚类中的注释准确率显著优于单维过滤的Lipid Data Analyzer(LDA)。在高分辨谱图再生测试中,WMYn模型在Orbitrap Exploris 240质谱系统的333例临床样本中表现卓越:低分辨率(两位小数)条件下以最优相似度领先均值法、线性拟合等方法;高分辨率(四位小数)时预测精度优势进一步扩大。整合五级谱库后,基于MS-DIAL数据库的注释实验在0.75相似度阈值下显著提升多数脂质亚类的Top-20召回率,验证了其在复杂脂质解析中的独特价值。
该软件针对学术用户开源,其良好的可扩展性使其成为一个极具潜力的工具。更多信息可以访问:https://github.com/LinShuhaiLAB/LipidIN, https://doi.org/10.5281/zenodo.14824498.

图2 LipidIN的质谱图库查询速度远超Flash Entropy
该研究由厦门大学医学院硕士生徐昊、生命科学学院硕士生姜天航、共享平台张蕾和福建医科大学附属协和医院林雨翔共同完成,我院林树海教授、宁波东方理工大学(暂名)蔡宗苇教授和集美大学曾珺教授为共同通讯作者。
聚焦线粒体中心磷脂(Cardiolipin)的结构解析难题,2025年3月3日,林树海教授团队在Chinese Chemical Letters上发表了题为Development of a comprehensive computational pipeline for cardiolipin atlas in an intermittent fasting model的研究论文,开发了线粒体中心磷脂的注释新算法,实现了对心磷脂复杂多样的结构式快速拼接。该计算框架还运用了间歇性禁食的动物组织中,绘制出多器官心磷脂图谱。更多代码信息可以访问:https://github.com/LinShuhaiLAB/CLAN.

该研究由厦门大学生命科学学院硕士生钟晓莉和陈亮生共同完成,我院林树海教授和集美大学曾珺教授为共同通讯作者。
2025年1月27日,林树海教授团队在Analytical Chemistry发表了题为QuanFormer: A Transformer-Based Precise Peak Detection and Quantification Tool in LC-MS-Based Metabolomics的研究论文。该研究基于transformer架构和计算机可视化方法,开发出准确识别液相色谱质谱峰并进行定量分析的端到端计算工具QuanFormer。不仅如此,对液相色谱中常见的保留时间漂移也有较好的校准效果。更多代码信息可以访问:https://github.com/LinShuhaiLAB/QuanFormer.

该研究由厦门大学人工智能研究院硕士生张正义和药学院硕士生杨欢共同完成,我院林树海教授为通讯作者。
原文链接:
https://www.nature.com/articles/s41467-025-59683-5
https://www.sciencedirect.com/science/article/pii/S100184172500213X
https://pubs.acs.org/doi/10.1021/acs.analchem.4c04531
(文/图 林树海教授团队)